前回Heart Disease(Cleveland)のデータ俯瞰を実施しました。
本記事では続きで下記を実施したいと思います。
- モデリング用データの準備
- モデリング
モデリングでは心臓病かどうかを当てるモデルを作成したいと思います。
色々候補はありますが、まずはロジスティック回帰でやってみたいと思います。
分析用データの作成
前回までに加工したデータを再現
欠損値処理と目的変数を作成しました。
import pandas as pd
column_names = [
'age', 'sex', 'cp', 'trestbps', 'chol', 'fbs', 'restecg',
'thalach', 'exang', 'oldpeak', 'slope', 'ca', 'thal', 'num'
]
df = pd.read_csv("/Users/hinomaruc/Desktop/blog/dataset/heart+disease/processed.cleveland.data", header=None, names=column_names)
# 数値型でない行の値をNaNにする
df['ca'] = pd.to_numeric(df['ca'], errors='coerce')
df['thal'] = pd.to_numeric(df['thal'], errors='coerce')
# 欠損値処理
ca_median = df.dropna().groupby("num")["ca"].median()
thal_median = df.dropna().groupby("num")["thal"].median()
df['ca'] = df['ca'].fillna(df['num'].map(ca_median))
df['thal'] = df['thal'].fillna(df['num'].map(thal_median))
# 目的変数の作成
df['target'] = df['num'].apply(lambda x: 1 if x >= 1 else 0)カテゴリ情報の変換について
Heart Disease(Cleveland)のデータセットは数値情報から構成されています。バイナリ情報や量を表現している場合はそのままでも良さそうですが、数値をカテゴリ情報として使っている場合は順序型の変数なのかどうかを判断する必要がありそうです。
順序型変数は、カテゴリ間に順序やランクが存在する変数です。具体的な例としては、悪い:1、普通:2、良い:3など高評価なほど数値が増えるなどでカテゴリを表現します。順序型変数はカテゴリを順序の情報を保持しつつ数値データとして扱える特徴があります。
順序型の変数でない場合はダミー変数化する必要があります。
ダミー変数は、カテゴリカルな変数をバイナリの数値変数として表現する手法です。順序やランクの情報は持ちませんが、カテゴリの存在を示すために使用されます。例えば、1月や2月など年の月の情報は1月が最低、12月が最高などと言った意味を持たない限りは順序に特段の意味を持たない変数かと思います。
(時系列データとして月次の周期性を考慮したい場合は除く)
この場合は月の情報をダミー変数化して機械学習の説明変数として使われることがあります。通常、n個のカテゴリがある場合、n-1個のダミー変数を作成します。理由はn番目のカテゴリは他のn-1個のダミー変数が全て0の場合に1であることが暗黙的に把握出来るからです。
月情報をダミー変数化した場合、1月を表現すると1月:1、2月~11月:0、12月:なし などで表現します。
なぜn-1個のダミー変数を作成するのかは多重共線性が関わって来ます。
例えば、性別を表すダミー変数を考えると、1つのダミー変数(例:男性を表すダミー変数)が0であれば、もう1つのダミー変数(例:女性を表すダミー変数)は必ず1になります。つまり、1つのダミー変数が他のダミー変数から完全に予測できる状態になります。(不明といった第三の選択肢もありますが、今回は考慮していません。) これにより、ダミー変数同士の間に高い相関が生じ、多重共線性の問題が発生する可能性があります。
多重共線性については過去記事にて少しだけまとめてありました。多重共線性が生じるとモデルが不安定になる場合があります。詳細はリンク先をご覧ください。
・[titanic] 多重共線性(multicollinearity)が存在することの影響
Heart Disease(Cleveland)のカテゴリ情報をダミー変数化する
さて、Heart Disease(Cleveland)のデータセットで数値がカテゴリ情報として使われているのは下記4つの変数になるのかなと思います。
- cp (1:典型的な狭心症、2:非典型的な狭心症、3:非狭心症痛、4:無症状)
- restecg (0: 正常、1:ST-T波異常を示す、2:左室肥大の可能性または明確な証拠がある)
- slope 1:上向き、2:平坦、3:下降)
- thal (3:正常、6:固定欠損、7:可逆性欠損)
正直どの変数も順序型の変数になり得るのか判断が難しかったので、今回は全てダミー変数として扱おうと思います。
例えば、thalは3区分(3:正常、6:固定欠損、7:可逆性欠損)存在しますが、固定欠損と可逆性欠損はどちらが症状が悪いのか専門家の力を借りないと判断は難しそうですし、cpに関しては事前のデータ俯瞰で4:無症状の方が心臓病の可能性が高そうという結果にあったので単純に順序型の変数として扱ってしまっていいのか決断することが出来ませんでした。
from sklearn.preprocessing import OneHotEncoder
# OneHotEncoderの定義 (各特徴量の最初の区分を落とす)
OneHotEnc = OneHotEncoder(categories='auto',drop='first',handle_unknown='ignore') #エラーは0になるオプション
# OneHotコンバート対象の変数
OneHotCols=["cp","restecg","slope","thal"]
# fit_transformして、ダミー変数の作成
get_dummies = OneHotEnc.fit_transform(df[OneHotCols])
# ダミー変数名取得
dummy_cols = OneHotEnc.get_feature_names_out()
# 元のデータフレームにダミー変数を追加する
df = df.join(pd.DataFrame(get_dummies.toarray(),columns=dummy_cols))
# ダミー化した変数を除外
df = df.drop(columns=OneHotCols)
# 作成データの確認
df.info()RangeIndex: 303 entries, 0 to 302 Data columns (total 20 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 age 303 non-null float64 1 sex 303 non-null float64 2 trestbps 303 non-null float64 3 chol 303 non-null float64 4 fbs 303 non-null float64 5 thalach 303 non-null float64 6 exang 303 non-null float64 7 oldpeak 303 non-null float64 8 ca 303 non-null float64 9 num 303 non-null int64 10 target 303 non-null int64 11 cp_2.0 303 non-null float64 12 cp_3.0 303 non-null float64 13 cp_4.0 303 non-null float64 14 restecg_1.0 303 non-null float64 15 restecg_2.0 303 non-null float64 16 slope_2.0 303 non-null float64 17 slope_3.0 303 non-null float64 18 thal_6.0 303 non-null float64 19 thal_7.0 303 non-null float64 dtypes: float64(18), int64(2) memory usage: 47.5 KB
ダミー変数化出来たようです。
# 相関係数確認 (r < 0.3は非表示)
import matplotlib.pyplot as plt
import seaborn as sns
plt.figure(figsize=(18,14))
corr=df.drop(columns=df.columns[[0]]).corr()
sns.heatmap(corr, vmax=1, vmin=-1, center=0, mask = abs(corr) < 0.3,linecolor="black",linewidth=0.5, annot=True,annot_kws={"size":8})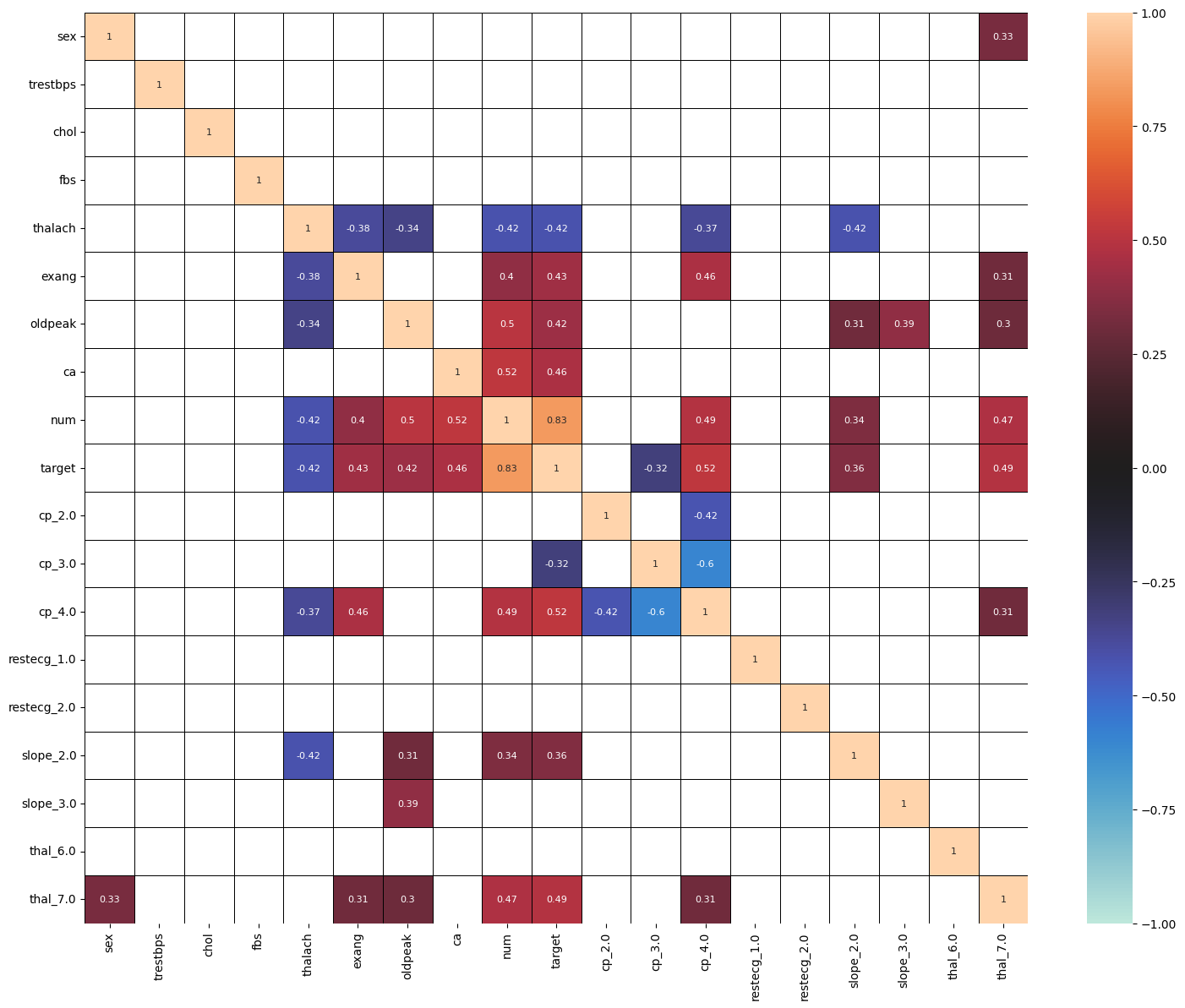
ロジスティック回帰で心臓病かどうかを当てるモデルを作成する
心臓病のリスクが高い人を当てるモデルと言った方が正しいのでしょうか。私が一番好きなロジスティック回帰を使ってモデリングしてみます。
df = df.drop(columns=["num"])
# 訓練データとテストデータに分割する。
from sklearn.model_selection import train_test_split
train, test = train_test_split(df, test_size=0.20,random_state=100)
# targetの割合を確認
test["target"].describe()count 61.000000 mean 0.442623 std 0.500819 min 0.000000 25% 0.000000 50% 0.000000 75% 1.000000 max 1.000000
44%のtargetが1なので、バランスは問題ないようですのでこのまま進めたいと思います。
本当は層別サンプリングなど実施した方がよりよい分割になると思います。男女の比率とかも気になるところ。
X_train = train.drop(columns=["target"]) # 説明変数 (train)
y_train = train["target"] # 目的変数 (train)
X_test = test.drop(columns=["target"]) # 説明変数 (test)
y_test = test["target"] # 目的変数 (test)
# https://scikit-learn.org/stable/modules/generated/sklearn.linear_model.LogisticRegressionCV.html
from sklearn.linear_model import LogisticRegressionCV
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import StandardScaler
# Cは正則化の度合いを表し、Csは最適なパラメータを探すためintを指定するとint分のCの値を作成してくれる。
pipeline = make_pipeline(StandardScaler(), LogisticRegressionCV(Cs=1000,cv=5,random_state=0))
# fitする
fit_pipeline = pipeline.fit(X_train,y_train)
# 訓練データへの当てはまりを確認
# Return the mean accuracy on the given test data and labels.
print(fit_pipeline.score(X_train,y_train))
# テストデータへの当てはまりを確認
# Return the mean accuracy on the given test data and labels.
print(fit_pipeline.score(X_test,y_test))0.8305785123966942 0.8852459016393442
訓練データへの当てはまり具合とテストデータへの当てはまり具合に差がそれほどないので、過学習はしていなさそうです。
Accuracy88.5%は中々良い精度ですね。
変数の寄与度の確認
どの変数がモデルに寄与しているのか出力してみます。心臓病かどうかを判別するのに重要な変数が何なのか確認します。
# pipelineだと係数(model.coef_)が確認できないので、pipelineからモデル部分を取り出す
model_pipeline = fit_pipeline.named_steps["logisticregressioncv"]
# 説明変数の係数を確認
coef = pd.DataFrame()
coef["features"] = fit_pipeline.feature_names_in_
coef["coef"] = model_pipeline.coef_[0]
coef["coef_pct"] = np.abs(coef["coef"]) / np.abs(coef["coef"]).sum()
coef.sort_values(by="coef_pct",ascending=False)| no | features | coef | coef_pct |
|---|---|---|---|
| 1 | cp_4.0 | 0.129580 | 0.116330 |
| 2 | thal_7.0 | 0.129028 | 0.115835 |
| 3 | ca | 0.115831 | 0.103987 |
| 4 | exang | 0.101836 | 0.091423 |
| 5 | thalach | -0.101183 | 0.090837 |
| 6 | oldpeak | 0.087890 | 0.078903 |
| 7 | slope_2.0 | 0.079952 | 0.071776 |
| 8 | cp_3.0 | -0.078302 | 0.070295 |
| 9 | sex | 0.066317 | 0.059536 |
| 10 | cp_2.0 | -0.042592 | 0.038236 |
| 11 | age | 0.042311 | 0.037985 |
| 12 | restecg_2.0 | 0.038834 | 0.034863 |
| 13 | trestbps | 0.030709 | 0.027569 |
| 14 | chol | 0.022775 | 0.020446 |
| 15 | thal_6.0 | 0.018729 | 0.016814 |
| 16 | restecg_1.0 | 0.017080 | 0.015333 |
| 17 | slope_3.0 | 0.010673 | 0.009581 |
| 18 | fbs | 0.000279 | 0.000250 |
cp,thal,ca,exang,thalachあたりが寄与しているようです。変数逆転現象も係数を確認する限り起きていなさそうですかね?
thalach(最大心拍数)はマイナスの係数ですが、こちらも事前にデータ俯瞰した結果と整合していると思うので問題ないと思います。(最大心拍数が低いほど心臓病のリスクが高かったと思います。)
まとめ
ロジスティック回帰だと88.5%の精度になりました。今回は全ての変数を投入したのですが、変数選択や特徴量エンジニアリングをして再モデリングをするなどやれることはありそうです。(精度あがるかは分かりませんが、、)
次はニューラルネットワークを使ってみようと思います。Kaggleにサンプルノートブックがありましたので参考にしつつやって見たいと思います。

